NEB全球首推甲基化领域的颠覆性技术革新:一种高效替代重亚硫酸盐转化的酶学转化法!
来源:非原创 2019-05-24 13:02
NEB全球首推甲基化领域的颠覆性技术革新:一种高效替代重亚硫酸盐转化的酶学转化法!DNA 甲基化图谱分析,尤其是在基因组中检测 5-甲基胞嘧啶(5mC)和5-羟甲基胞嘧啶(5hmC),是至关重要的,甲基化修饰会影响基因的表达。通常,转录起始位点附近的低甲基化水平与较高的转录水平有关,而调节区域内高水平胞嘧啶修饰的基因则出现较低转录水平。完整和准确的甲基化图谱分析在许多领域都很重要:包括癌症等疾病的
NEB全球首推甲基化领域的颠覆性技术革新:一种高效替代重亚硫酸盐转化的酶学转化法!

DNA 甲基化图谱分析,尤其是在基因组中检测 5-甲基胞嘧啶(5mC)和5-羟甲基胞嘧啶(5hmC),是至关重要的,甲基化修饰会影响基因的表达。通常,转录起始位点附近的低甲基化水平与较高的转录水平有关,而调节区域内高水平胞嘧啶修饰的基因则出现较低转录水平。完整和准确的甲基化图谱分析在许多领域都很重要:包括癌症等疾病的研究、胚胎发育监测和农业植物研究。然而,现有的甲基化图谱分析技术存在明显的缺陷。
全基因组重亚硫酸盐测序(WGBS)长期以来一直是甲基化图谱分析的金标准,但重亚硫酸盐的化学反应会破坏和降解 DNA,导致 DNA 断裂和丢失。此外,重亚硫酸盐文库表现出明显的 GC 偏嗜,并在甲基化区域尤为显著。
为了克服这些局限性,NEB 开发了一种基于酶学转化、能同时检测 5mC 和5hmC 的新方法——NEBNext 酶学转化法甲基化建库试剂盒 (EM-seq)。
高效的酶法转化可最大程度地减少对 DNA 的损伤,结合提供的 NEBNext Ultra™ II 文库制备流程,最终产生的高质量文库可以从有限的测序数据中更灵敏的检测到 5mC 和 5hmC。EM-Seq方法得到的结果与 WGBS 得到的转化序列相同,因此可以使用相同的数据分析流程。
工作流程
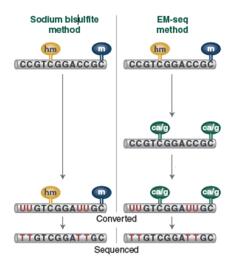
EM-Seq 流程的第一步,与 WGBS 方法相同,都是先从打断 DNA 开始构建文库。EM-Seq 的标准起始量为 10-200 ng 的打断后的 DNA,优化后的流程也可用于高达 500 ng 的起始量。紧接着是两步酶法转化,目的是将未甲基化的胞嘧啶与 5mC 和 5hmC 区分开来。最后,在上机测序前对文库进行 PCR 扩增。Em-seq 的两步酶促转化与重亚硫酸盐转化如图 1 所示。
图1:EM-Seq 和重亚硫酸盐转化的方法原理
结果展示
1. 更长的文库插入片段
相较于过于损伤 DNA 的重亚硫酸盐处理,EM-seq 流程对 DNA 更为温和,最大程度地减少了对 DNA 的损伤。结果显示,EM-seq 转化后的 DNA 比重亚硫酸盐转化后的 DNA 更为完整,得到的文库中拥有更多更长的插入片段,如图 2 所示。最终能获得更长的序列和更高的比对率,同时尽可能地降低测序成本。
图 2:NEBNext 酶学转化法甲基化文库可获得更长的插入片段

采用 Covaris® S2 仪器将 50 ng 人 NA12878 基因组 DNA 打断至 300 bp,同时作为 EM-Seq 和 WGBS 建库的起始样本。对于 WGBS 方法,使用 NEBNext Ultra II DNA 进行建库,随后采用 Zymo Research EZ DNA Methylation-Gold™ 试剂盒进行重亚硫酸盐转化。两个文库均使用 Illumina MiSeq(2 X 76 bases)测序,片段大小采用 Picard 2.18.14 测定。图中绘制了每个插入片段出现的频率标准化后的数据,结果如图:EM-Seq 建库后的插入片段比 WGBS 方法得到的插入片段更长,说明:酶学转化法不会对 DNA 造成损伤,而重亚硫酸盐处理的 DNA 有严重损伤。
2. 更高的文库产量
重亚硫酸盐转化引起的 DNA 损伤、断裂和丢失降低了其文库扩增后的产量。相反,在 EM-seq 流程中更温和的转化方式可保证获得高质量的 DNA 文库,继而进行高效扩增。因此,相较于 WGBS,EM-seq 可用更少的 PCR 循环实现更高的文库产量(图3)。
图3:相较于 WGBS,EM-Seq 能在更少的 PCR 循环下得到更高的文库产量
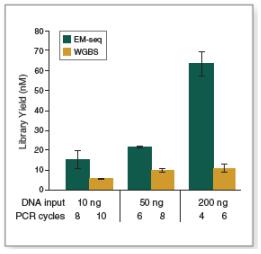
采用 Covaris S2 仪器将 10 ng、50 ng 和 200 ng 不同起始量的人 NA12878 基因组 DNA 打断至 300 bp,同时作为 EM-Seq 和 WGBS 建库的起始样本。对于 WGBS 方法,使用 NEBNext Ultra II DNA 进行建库,随后采用 Zymo Research EZ DNA Methylation-Gold 试剂盒进行重亚硫酸盐转化。上述所有起始量,EM-Seq 都能使用更少的 PCR 循环得到更高的文库产量,表明 EM-Seq 显著减少了 WGBS 方法中的 DNA 损失。误差条表示标准差。
3. 更均一的 GC 覆盖度
虽然文库产量是测序成功的要素,但文库质量也同样至关重要。一个高质量文库应能真实反映原始样本情况,包括均一的 GC 覆盖度。
由于重亚硫酸盐转化时会损伤非甲基化胞嘧啶,而非甲基化胞嘧啶占所有胞嘧啶的绝大部分,因此这种导致损伤的转化方式会对 GC 比例造成影响。这种特定的损伤、断裂和丢失导致重亚硫酸盐转化后的文库在 GC 测序不足,而在 AT 过度测序。相比之下,EM-seq 文库显示了更均一的 GC 覆盖度,结果表明:EM-seq 对 DNA 的损伤更少,更能真实反映原始样本的情况。(图 4)。
图4:EM-Seq能实现更卓越的 GC 均一覆盖度。
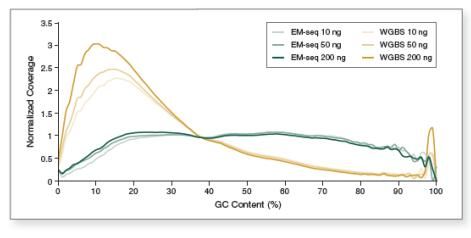
采用 Covaris S2 仪器将 10 ng、50 ng 和 200 ng 不同起始量的人 NA12878 基因组 DNA 打断至 300 bp,同时作为 EM-Seq 和 WGBS 建库的起始样本。对于 WGBS 方法,使用 NEBNext Ultra II DNA 进行建库,随后采用 Zymo Research EZ DNA Methylation-Gold™ 试剂盒进行重亚硫酸盐转化。两个文库均使用 Illumina NovaSeq® 6000(2 X 100 bases)测序,使用 bwa-meth 0.2.2 将测序数据与 hg38 进行比对。使用 Picard 2.17.2 计算 GC 覆盖度,图中显示不同 GC 含量时(0-100%),标准化后覆盖度的分布情况。结果显示:EM-Seq 文库显著提高 GC 覆盖度的均一性,无 AT 过度测序,也无 GC 测序不足,后两项都是 WGBS 文库的典型缺陷。
4. 更高的CpG 位点检测灵敏度
分析两种转化方法在不同测序深度下检测到的 CpG 位点数量,可以比较 EM-seq 和 WGBS 对 CpG 位点的检测灵敏度。图 5 显示:在 10 ng、50 ng 和 200 ng 三种起始量下,分析相同的数据量(3.24 亿),EM-seq 文库能比重亚硫酸盐文库检测到更多的 CpG 位点,并且在更低测序深度下,EM-Seq 也能检测到更多的 CpG 位点。例如,用 EM-Seq 和 WGBS 两种方法在不同起始量情况下,至少 1X 和 8X 测序深度下检测到的独有和共有的 CpG 位点数量,EM-Seq 在至少 1X 测序深度下比 WGBS 多检测到 20% 以上的 CpG 位点。而在至少 8X 测序深度下,CpG 位点覆盖度差异增加至 2 倍。
图5:相较于 WGBS,EM-Seq 在更低测序深度能检测到更多的 CpG 位点。
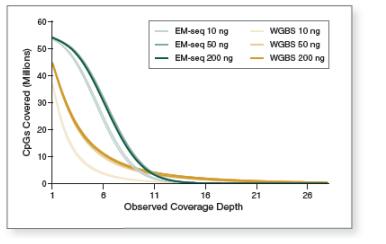
使用 bwa-meth 0.2.2 将 测序数据 与 hg38 进行比对。通过分析 3.24 亿双端数据得到 EM-Seq 和 WGBS 文库的 CpG 位点覆盖度,其中每条链都独立计数,最终得到最多 5600 万个可能的 CpG 位点。结果显示:EM-Seq 在更低测序深度能检测到更多的 CpG 位点。
图 6 显示了 EM-Seq 和 WGBS 两种方法在不同起始量,不同测序深度,采用相同测序数据检测到的独有和共有的 CpG 位点,同样表明 EM-seq 能够检测到更多的 CpG 位点,并且这种差异在提高测序深度后更为显著(图 6)。在至少 1X 测序深度下,使用 10、50 和 200 ng 作为起始样本,EM-seq 能检测到约 5400万个 CpG 位点,但 WGBS 只识别了约 3600 万至 4600 万个 CpG 位点。在至少 8X 测序深度下,EM-seq 检测到的 CpG 位点数在 1100 万至 1660 万,而 WGBS 仅检测到 160 万至 710 万。结果表明,与 WGBS 相比,EM-seq 能从更少的数据量中挖掘到更多的相关信息(CpG 覆盖度)。
图6:相较于 WGBS,EM-Seq 在更低的测序深度情况下能检测到更多的 CpG 位点
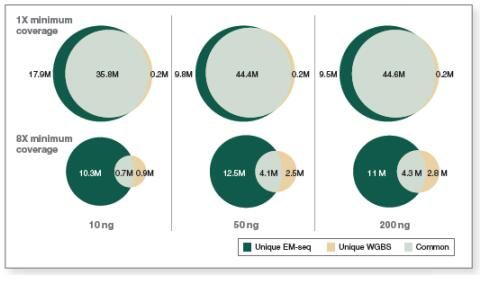
采用 Covaris S2 仪器将 10 ng、50 ng 和 200 ng 不同起始量的人 NA12878 基因组 DNA 打断至 300 bp,同时作为 EM-Seq 和 WGBS 建库的起始样本。对于 WGBS 方法,使用 NEBNext Ultra II DNA 进行建库,随后采用 Zymo Research EZ DNA Methylation-Gold™ 试剂盒进行重亚硫酸盐转化。两个文库均使用 Illumina NovaSeq® 6000(2 X 100 bases)测序,使用 bwa-meth 0.2.2 将测序数据与 hg38 进行比对。通过分析 3.24 亿双端数据得到 EM-Seq 和 WGBS 文库的 CpG 位点覆盖度。
图中显示了 EM-Seq 和 WGBS 两种方法在不同起始量,至少 1X 和 8X 测序深度下检测到的独有和共有的 CpG 位点。EM-Seq 在至少 1X 测序深度下比 WGBS 多检测到 20% 以上的 CpG 位点。而在至少 8X 测序深度下,CpG 位点覆盖度差异增加至 2 倍。
5. 更高的一致性
不同起始量和不同方法所产生的文库相关性,是评估一致性和高性能的有效参数。图 7 显示了采用 EM-seq 和 WGBS 两种方法,在不同起始量下,检测了 12 个文库各 2100 万个 CpG 位点间的 CpG 甲基化相关性。EM-seq 文库在不同起始量之间的相关性都很高,表明甲基化检测灵敏度不会随着起始量的减少而降低。相比之下,WGBS 文库之间的相关性最低。只有在较高的起始量下,EM-seq 和 WGBS 文库之间的相关性才最高。综上所述,表明 EM-seq 具有更高的一致性。
图 7:相较于 WGBS,EM-Seq 文库具有更高的 CpG 相关性
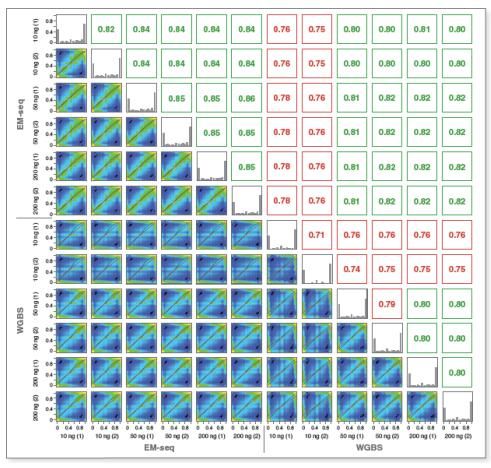
采用 methyKit 绘制 10、50 和 200 ng 起始量下 EM-seq 和 WGBS 文库之间的相关性,最小覆盖度为 1×(所有文库均使用 2100 万个 CpG 位点)。EM-seq 文库在不同起始量之间的相关性都很高,Pearson 相关系数在 0.82 到 0.86 之间。而 WGBS 文库之间的相关性仅在 0.71 到 0.8 之间。起始量在 50 ng 和 100 ng 时,EM-seq 和 WGBS 之间的相关性最高,而在起始量降至 10 ng 时,两者的相关性最低。结果表明:比较 EM-seq 和 WGBS 数据后,发现 EM-seq 具有更高的一致性。
6. 更精准地检测转录起始位点的甲基化数据
在基因表达中,转录起始位点区域(TSS)通常是非甲基化的,而在基因较低水平表达时,调控区域的胞嘧啶甲基化水平较高。因此,能够准确检测转录调控元件是至关重要的。然而,如上文所述,WGBS 数据始终具有较低的含 C 二核苷酸表达问题,最终可能导致重要转录调控元件周围的 CpG 数据丢失。
图 8 显示了转录起始位点(TSS)周围的 CpG 覆盖度和甲基化情况。在 TSS 周围 4 kb 区域中,EM-seq 得到的 CpG 覆盖度显著高于 WGBS,并且更均一,也没有在 TSS 处出现 CpG 覆盖度下降的低峰,而 WGBS 在 TSS 处表现明显低峰。在 8X 条件下,EM-seq 获得的甲基化信息也与预期的甲基化模式更为一致,越接近 TSS 区域,甲基化水平越低。
图 8:EM-seq在检测转录起始位点周围的 CpG 甲基化方面优于 WGBS
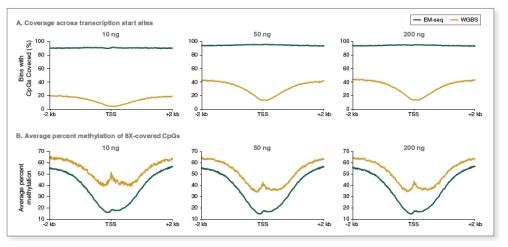
图中检测了不同起始量下转录起始位点(TSS)周围 CpG 位点甲基化情况。TSS 周围的 4 kb 被分成了 400 个 10 bp,在该区域内 CpG 位点被覆盖 8× 及以上的才能被用来评估甲基化水平。A:结果表示:EM-seq 在 TSS 区域有更高且更均一的覆盖度。B:图中显示了 EM-seq 和 WGBS 在 8× 覆盖度下,CpG 位点的平均甲基化百分比。 EM-seq 在 TSS 区域的数据更能代表预期的甲基化模式:TSS 区域的甲基化水平最低,而在+/-2 kb 处甲基化水平增高。
虽然重亚硫酸盐测序是研究 DNA 甲基化的金标准,但这种转化方式会对DNA 造成损伤,导致 DNA 断裂、丢失和 GC 偏嗜。NEBNext 酶学转化法甲基化建库试剂盒提供了一种酶学方法代替全基因组重亚硫酸盐处理 DNA (WGBS) 的方法,并结合高效流程化的建库步骤,适用于 Illumina 平台测序。
高效的 EM-Seq 酶促转化可最大程度地减少对DNA的损伤,结合提供的 NEBNext Ultra II 文库制备流程,最终制备的高质量文库可以从有限的测序数据中更灵敏的检测到 5-mC 和 5-hmC。
优势
更卓越的 5-mC 和 5-hmC 检测灵敏度
更高的比对率
更均一的 GC 覆盖度
能从有限的测序数据检测到更多的 CpG 位点
更均一的二核苷酸分布
更长的文库插入片段
更高效的建库流程
提供转化模块
扫描下方二维码可申请参与“NEB 甲基化测序建库体验星标计划”。

如需产品订购请联系您身边的 NEB 授权经销商或拨打技术支持热线 400-811-2220 / 400-690-3366

版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


